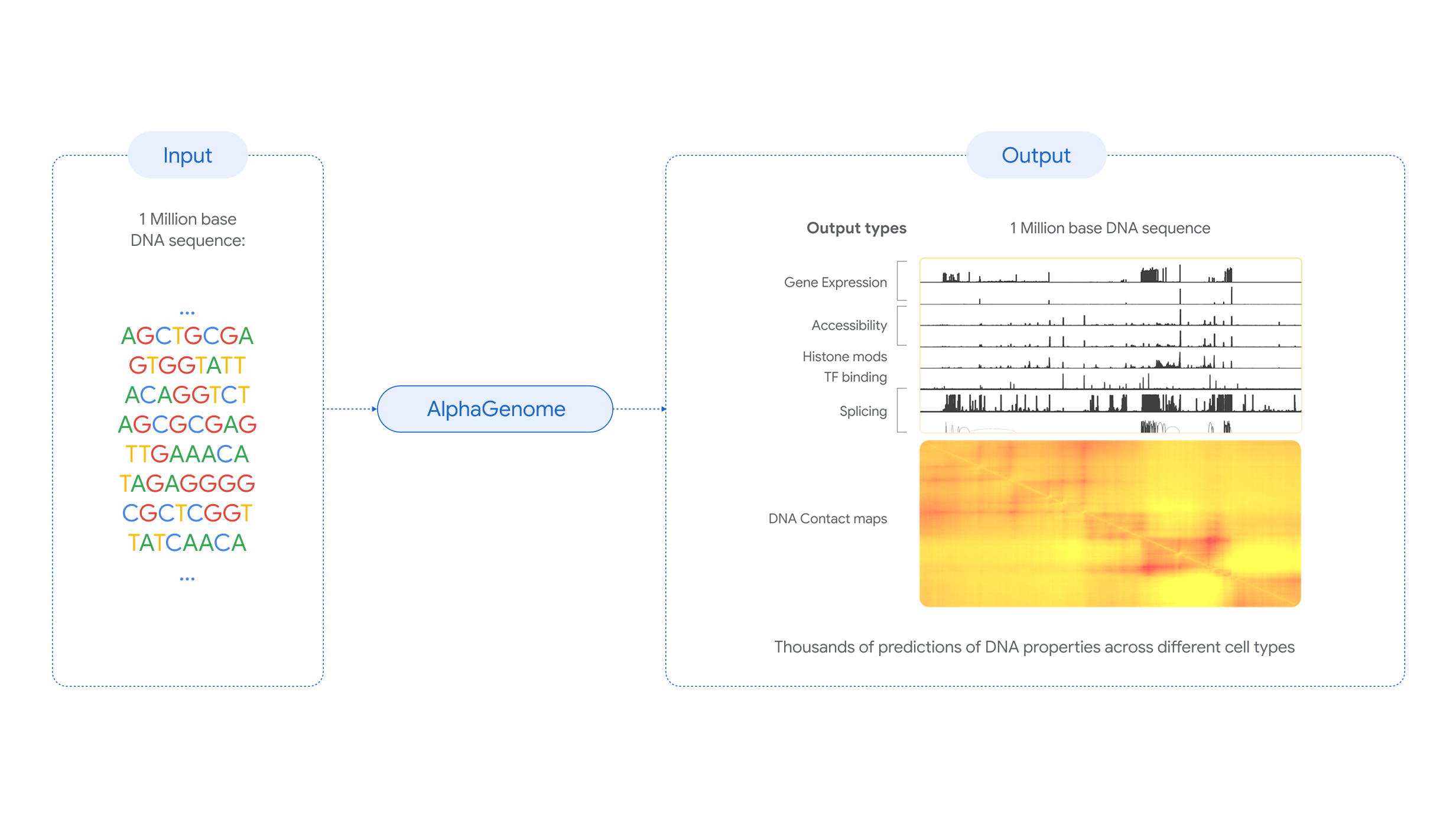
- AlphaGenome analiza secuencias de ADN de hasta un millón de letras y se centra en el ADN no codificante.
- La IA de Google DeepMind predice el impacto de variaciones genéticas en la regulación de los genes y en la aparición de enfermedades.
- El modelo iguala o supera a casi todas las herramientas previas en 25 de 26 pruebas estándar publicadas en Nature.
- Investigadores europeos destacan su potencial para mejorar diagnósticos genéticos y el desarrollo de terapias, aunque recuerdan sus límites y la dependencia de buenos datos.
La irrupción de AlphaGenome, la nueva inteligencia artificial de Google DeepMind, está dando un giro importante a la forma en que los científicos estudian el genoma humano. Este modelo de aprendizaje profundo se ha diseñado para predecir cómo influyen las variaciones en el ADN en el funcionamiento de los genes y en los procesos biológicos que regulan la vida.
Lejos de ser solo otro avance llamativo en inteligencia artificial, AlphaGenome se coloca en el centro de uno de los grandes retos de la biología moderna: interpretar la enorme parte del genoma que no codifica proteínas pero que controla cuándo, dónde y con qué intensidad se activan los genes. Comprender estas regiones promete mejorar el diagnóstico genético y orientar el desarrollo de nuevas terapias, especialmente en Europa, donde la investigación en genómica y medicina de precisión está ganando cada vez más peso.
Del número de genes a la «gramática» del genoma
A primera vista puede sorprender que un ratón, un rodaballo y una persona tengan aproximadamente el mismo número de genes, en torno a 20.000, y sin embargo sean organismos tan distintos. El motivo es que el simple recuento de genes dice muy poco sobre la complejidad de un organismo o sobre cómo va a desarrollarse.
La clave está en que esos 20.000 genes solo ocupan alrededor del 2 % del genoma. El 98 % restante, que durante años se llegó a llamar «ADN basura», incluye multitud de secuencias repetitivas, elementos móviles como transposones y retrotransposones, y, sobre todo, regiones reguladoras que indican a cada gen cuándo encenderse y apagarse, en qué tejidos y en qué fase del desarrollo.
En otras palabras, la diversidad entre especies no se logra sumando o restando genes, sino modificando el programa que controla su activación y silenciamiento. Cambiar estas instrucciones reguladoras con los mismos genes de base puede dar lugar a organismos completamente diferentes, algo que ayuda a explicar por qué un humano, un pez plano o un roedor pueden compartir un número similar de genes y, sin embargo, tener biologías tan dispares.
Durante años se han utilizado algoritmos informáticos para rastrear esa parte no codificante del genoma en busca de señales reguladoras concretas. Muchos programas se especializaban en un solo tipo de elemento: activadores, silenciadores o aisladores que separan el efecto regulador entre genes vecinos. Estas herramientas comparaban secuencias problema con patrones conocidos, pero normalmente sobre tramos relativamente cortos de ADN, de unos pocos miles de nucleótidos.
AlphaGenome rompe con ese enfoque fragmentado. Según el trabajo publicado en la revista Nature, esta IA es capaz de analizar de una sola vez segmentos de ADN de hasta un millón de letras y de predecir simultáneamente el comportamiento de múltiples tipos de elementos reguladores, lo que supone un salto en escala y en capacidad de integración respecto a lo que se hacía hasta ahora.
Qué es AlphaGenome y qué tiene de distinto
AlphaGenome es un modelo de inteligencia artificial de aprendizaje profundo desarrollado por Google DeepMind que se ha entrenado con datos del genoma humano y del ratón. Su objetivo central es predecir cómo las variaciones en la secuencia de ADN afectan a los procesos que regulan los genes, con especial énfasis en el ADN no codificante.
La herramienta ha sido evaluada en 26 pruebas estándar frente a otros modelos existentes y, según los datos difundidos en Nature, igualó o superó su capacidad de predicción en 25 de esas 26 comparativas. Una de sus mayores ventajas es que puede trabajar con fragmentos largos de código genético y, aun así, ofrecer una resolución muy fina a nivel de nucleótidos individuales.
En términos prácticos, esto significa que AlphaGenome puede predecir la función biológica de secuencias extensas de ADN y anticipar cómo determinadas mutaciones pueden alterar la expresión génica, la producción de ARN o el patrón de encendido y apagado de los genes. Esta información resulta especialmente valiosa en aquellas zonas del genoma que se habían pasado por alto en muchos análisis clínicos y de investigación.
Los autores del modelo, entre ellos Žiga Avsec, Natasha Latysheva y Pushmeet Kohli, explican que la IA se ha alimentado de grandes colecciones de datos públicos sobre ADN no codificante en numerosos tipos de células y tejidos humanos y de ratón. De este modo, el sistema ha podido aprender las «reglas» de la regulación genética en distintos contextos biológicos.
Google DeepMind presenta esta tecnología como una herramienta orientada a convertir el código estático del genoma en un lenguaje interpretable. Si el ADN fuera un libro, el proyecto aspira a descifrar su gramática: qué combinaciones de letras regulan la actividad de los genes, de qué depende que una variante se asocie a una enfermedad o pase desapercibida y cómo se coordinan las señales que actúan a largas distancias dentro del genoma.
El foco en el ADN no codificante y la «materia oscura» del genoma
Desde que se completó el mapa del genoma humano, allá por 2003, se supo que la inmensa mayoría del ADN no codifica proteínas. Durante un tiempo se calificó buena parte de esas regiones como «ADN basura», precisamente porque no se entendía su función.
Con el tiempo, los estudios han ido demostrando que ese 98 % del genoma desempeña un papel decisivo en la regulación de la actividad genética. Estas zonas determinan qué genes se activan, en qué tipo de célula, en qué momento del desarrollo y con qué intensidad. En ellas se concentra, además, la mayor parte de las variantes genéticas asociadas a enfermedades complejas.
AlphaGenome se ha diseñado expresamente para interpretar esta «materia oscura» del genoma. Su capacidad para procesar tramos de hasta un millón de pares de bases le permite tener en cuenta señales reguladoras que pueden encontrarse a gran distancia del gen al que afectan, algo que los modelos más antiguos, limitados a fragmentos cortos, solo podían aproximar de manera parcial.
Este enfoque resulta especialmente relevante en campos como la oncología, la cardiología o la neurología, donde pequeñas modificaciones en regiones reguladoras pueden desencadenar cánceres, enfermedades cardiovasculares, trastornos neurológicos o síndromes genéticos poco frecuentes. Muchas de estas variantes no alteran directamente la secuencia de un gen, sino que cambian el contexto regulador en el que ese gen se activa o se silencia.
Para los equipos europeos que trabajan en medicina de precisión, disponer de una IA que ayude a evaluar el efecto de cambios fuera de los genes clásicos supone abrir la puerta a reinterpretar pruebas genéticas que antes se consideraban negativas o poco concluyentes. Variantes que se encontraban alejadas de los exones, por ejemplo, podrían adquirir un nuevo significado clínico a la luz de estos modelos.

Predicción del impacto de las mutaciones genéticas
Uno de los puntos fuertes de AlphaGenome es su capacidad para predecir cómo afectan las mutaciones genéticas a los procesos que regulan los genes. En las pruebas recogidas por Nature, el modelo ha mostrado un alto nivel de precisión a la hora de anticipar qué cambios en la secuencia pueden alterar la expresión génica o interrumpir procesos clave como el empalme del ARN.
Los investigadores de DeepMind subrayan que la herramienta puede evaluar simultáneamente miles de señales genéticas asociadas al funcionamiento de los genes y de las proteínas. Esto permite comparar un segmento de ADN «normal» con su versión mutada y estimar, de forma computacional, cuál será el efecto de esa alteración en la biología de la célula.
Este tipo de análisis es crucial en el estudio de enfermedades raras, trastornos complejos o casos en los que no se encuentra una mutación clara dentro del gen sospechoso. En muchos pacientes, la alteración puede hallarse a cierta distancia del gen, en una región reguladora que controla su encendido o apagado. Hasta ahora, identificar esas variantes y probar su relevancia funcional era costoso y lento.
Los autores del trabajo destacan que AlphaGenome no sustituye a los experimentos de laboratorio, pero sí puede ayudar a priorizar qué variantes conviene estudiar de forma experimental. Al filtrar de antemano millones de posibles cambios, los investigadores pueden concentrar recursos en las alteraciones que la IA señale como más probables de tener impacto biológico.
Para los laboratorios clínicos europeos, esto se traduce en posibilidades de afinar los diagnósticos genéticos y en una mejor interpretación de los informes de secuenciación, tanto en hospitales públicos como en centros de referencia en genómica repartidos por España y el resto del continente.
Una herramienta con aspiraciones clínicas y de investigación
Los responsables de DeepMind apuntan que AlphaGenome está ya disponible para su uso con fines de investigación no comercial y que son miles los científicos que han comenzado a integrarlo en sus trabajos, desde estudios básicos sobre regulación génica hasta proyectos más aplicados en biomedicina.
La compañía insiste en que esta IA puede impulsar nuevos descubrimientos biológicos y contribuir al desarrollo de terapias más ajustadas al perfil genético de cada persona. El modelo se presenta como una pieza más dentro del ecosistema de herramientas que buscan acelerar la transición hacia una medicina más personalizada.
En Europa, instituciones como el Instituto Francis Crick de Londres o el Instituto Wellcome Sanger de Cambridge han participado activamente en la evaluación independiente de la herramienta. Sus equipos han sometido AlphaGenome a centenares de miles de nuevos experimentos para verificar hasta qué punto las predicciones del modelo se sostienen en contextos distintos a los usados durante el entrenamiento.
Según los datos compartidos por estos centros, el rendimiento general de la IA ha sido muy elevado, especialmente en tareas que parecen seguir reglas «gramaticales» relativamente rígidas en el ADN, como la predicción de sitios de empalme. En estos casos, los expertos consideran que AlphaGenome podría sustituir sin grandes problemas a herramientas más antiguas que se han usado durante años en los análisis genéticos.
La posibilidad de estimar la expresión génica a partir de la sola secuencia de ADN se perfila como otro de los aspectos más prometedores del sistema. Aunque la vida real es más compleja y también intervienen factores ambientales, epigenéticos y de estilo de vida, disponer de un punto de partida fiable desde la secuencia facilita mucho el trabajo de investigadores y clínicos.

La visión de la comunidad científica europea
Varios expertos europeos han valorado el lanzamiento de AlphaGenome como un avance relevante en la intersección entre inteligencia artificial y genómica, aunque mantienen cierto grado de prudencia respecto a sus limitaciones actuales.
Desde el Centro Nacional de Biotecnología (CNB-CSIC), en España, el genetista Lluís Montoliu señala que esta herramienta tendrá un impacto significativo en la investigación básica, al ayudar a desentrañar cómo funcionan los genes y sus elementos reguladores. Al mismo tiempo, destaca que su utilidad se extiende a ámbitos más prácticos, como la mejora de los diagnósticos genéticos en contextos clínicos.
Montoliu insiste en que AlphaGenome permitirá interpretar regiones que hasta hace poco se consideraban «sin sentido» del genoma humano. Esa información gana importancia cuando se analizan casos donde no se detecta ninguna mutación evidente en un gen, pero el paciente presenta síntomas compatibles con una patología de base genética.
En Reino Unido, Robert Goldstone, responsable de Genómica en el Instituto Francis Crick, considera que AlphaGenome representa un hito importante en la inteligencia artificial aplicada al estudio del genoma. A su juicio, el modelo lleva la tecnología «del interés teórico a la utilidad práctica», ya que permite simular de forma programática las raíces genéticas de enfermedades complejas.
Goldstone matiza, no obstante, que la herramienta no es una solución mágica a todas las cuestiones biológicas. La expresión génica está profundamente influida por el entorno celular y por factores ambientales que la IA no puede observar directamente. Lograr el nivel de precisión actual basándose solo en reglas locales del ADN ya es una proeza técnica, pero aún queda margen para mejorar.
Potencial, límites y próximos pasos
Ben Lehner, director de Genómica Generativa y Sintética en el Instituto Wellcome Sanger de Cambridge, subraya que AlphaGenome es un ejemplo notable de cómo la IA está acelerando el descubrimiento biológico y el diseño de nuevas terapias. En sus pruebas, el modelo ha mostrado un rendimiento sólido incluso cuando se ha enfrentado a más de medio millón de experimentos adicionales no usados durante el entrenamiento.
Para Lehner, una de las grandes aportaciones de la herramienta será identificar diferencias específicas en nuestros genomas que aumentan o reducen la probabilidad de desarrollar enfermedades. Esa información es clave para diseñar fármacos más precisos y estrategias preventivas adaptadas a grupos de riesgo concretos.
Al mismo tiempo, el investigador recuerda que la calidad de cualquier modelo de IA depende en gran medida de los datos con los que se entrena. En biología, muchos conjuntos de datos son todavía pequeños, heterogéneos y poco estandarizados, lo que limita la capacidad de las máquinas para generalizar a todos los contextos posibles.
El reto ahora, según los expertos, es generar datos biológicos más amplios, fiables y comparables que permitan entrenar la próxima generación de modelos aún más potentes. Además, reclaman que tanto esos datos como las herramientas derivadas estén disponibles de manera abierta para toda la comunidad científica, también en Europa y España, donde numerosos grupos de investigación podrían beneficiarse de este tipo de recursos.
AlphaGenome se suma así a la estela de otros proyectos de DeepMind como AlphaFold, la IA que revolucionó la predicción de la estructura tridimensional de las proteínas. Si aquella iniciativa ayudó a entender cómo se pliegan las proteínas, este nuevo modelo aspira a desentrañar cómo funciona el genoma en su conjunto, con especial énfasis en la parte no codificante que durante tanto tiempo se dejó de lado.
Con todo, el escenario que se dibuja es el de una biología cada vez más apoyada en modelos computacionales capaces de leer y reinterpretar el código genético a gran escala. Herramientas como AlphaGenome no sustituyen a la experimentación, pero sí permiten ahorrar tiempo, orientar mejor las hipótesis y sacar más partido a la enorme cantidad de información genómica disponible; un cambio de paso que, si se consolida, puede traducirse en diagnósticos más finos, terapias mejor dirigidas y una comprensión mucho más detallada de cómo pequeñas variaciones en el ADN pueden llegar a condicionar nuestra salud.
